南科大生物系吴柘课题组揭示植物基因表达在染色质水平调控的新机制
日期:2019-12-26
近日,我校生物系、南科大-北大植物与食品研究所助理教授吴柘课题组与南京农业大学吴玉峰课题组在Molecular Plant(《分子植物》)(IF:10.8)在线发表研究论文,首次在模式植物拟南芥中系统研究了转录与RNA剪接的偶联过程,初步揭示了植物共转录RNA剪接的规律及调控机制。
RNA剪接是真核生物遗传信息解码中的关键步骤。在过去30年中,人们对酵母和动物细胞中RNA剪接的机制有了深入的认识,但对植物RNA剪接的机制仍了解有限。虽然剪接复合体从结构到功能在酵母和人类中是大致保守的,但不同物种在剪接调控机制上仍然可能存在极大的差异。例如,植物基因的内含子在哺乳动物细胞中通常不能正常剪接,反之亦然。
除了可变剪接之外,剪接的另一个重要特性和调控在于和转录过程的偶联,即边转录边剪接(或共转录剪接)。转录不仅仅在于生成RNA,也在于转录同时对RNA分子进行分选、加工、标记,初步确定RNA的命运(输出、翻译、降解等)。剪接也可以看作是RNA命运决定机制的一部分。
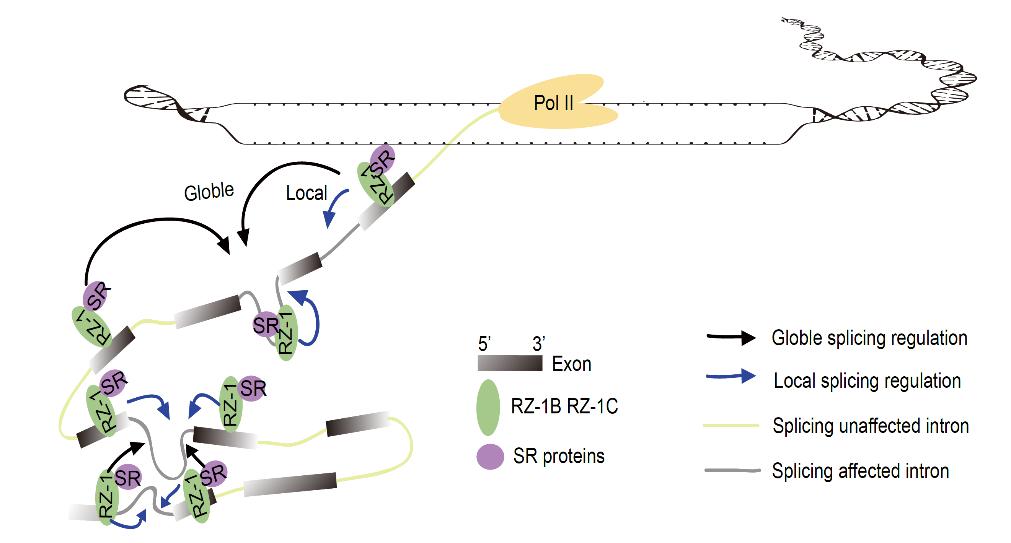
剪接因子-外显子-内含子间可能通过高维度的互作共同促进高效的共转录剪接
此前来自酵母、果蝇及哺乳动物细胞中的数据表明,在这些物种中共转录剪接普遍存在,且共转录剪接的效率与转录速率,基因结构,RNA聚合酶CTD上的磷酸化修饰等都有关联。然而遗憾的是,植物中相应的调控机制一直没有系统的研究报道。
利用模式植物拟南芥为研究对象,通过对染色质结合的RNA的纯化及测序分析(CB-RNA-seq),研究团队的工作显示共转录剪接在目前已检测的物种中是普遍模式。同时,植物的共转录剪接存在几个重要的特征:首先, 共转录剪接的效率与特定内含子到基因3’端的距离正相关,与一些激活型的组蛋白修饰(H3K4me3/H3K9ac)负相关,暗示快速的转录起始及延伸不利于共转录剪接,这与之前动物中提出的“动态耦合模型”一致;第二,mRNA中可变剪接的模式大致与染色质结合的RNA(CB-RNA)上保持一致,暗示可变剪接事件很大程度上是在染色质层面决定的;第三,此前在其它物种中并未关注到的是,一个基因共转录剪接的效率与它包含的内含子个数相关,而与基因长度不相关,暗示不同外显子/内含子的剪接事件之间存在相互促进协同的关系。
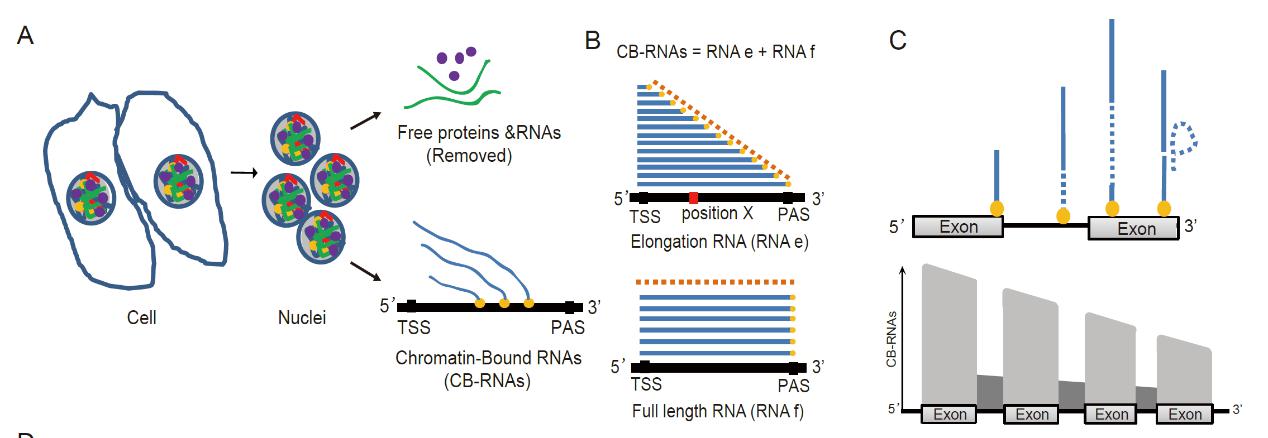
A:CB-RNA的来源及提取过程;B:CB-RNA对应由两种组分的RNA构成,其中RNAe代表正在转录延伸的RNA,RNAf代表转录延伸结束但是尚未从染色质上脱离的RNA;C: 假设RNA被共转录剪接的前提下,CB-RNA延染色质分布的预期情况示意
团队还研究了拟南芥两个hnRNP蛋白RZ-1B及RZ-1C对共转录剪接的调控机制。RZ-1B/1C此前被报道能够紧密结合染色质并且与多个剪接调控因子SR蛋白存在互作。团队发现,RZ-1B/1C对于共转录剪接的效率有全局性的增强作用。通过建立植物中的增强型的紫外交联结合免疫沉淀测序技术(eCLIP-seq),作者定位了体内RZ-1C与RNA底物分子的精确结合位点图谱,发现RZ-1C对共转录剪接的促进作用与它对这些RNA的直接结合是整合在一起的。有趣的是,RZ-1C主要结合在外显子上,且这种特点并不是由于内含子在细胞内稳态丰度低造成的。据此,作者提出外显子或者内含子可能作为剪接因子(RNA结合蛋白)的结合位点,同时借助外显子, 内含子与剪接因子间可能存在的高维度互作,共同促进高效的共转录剪接。
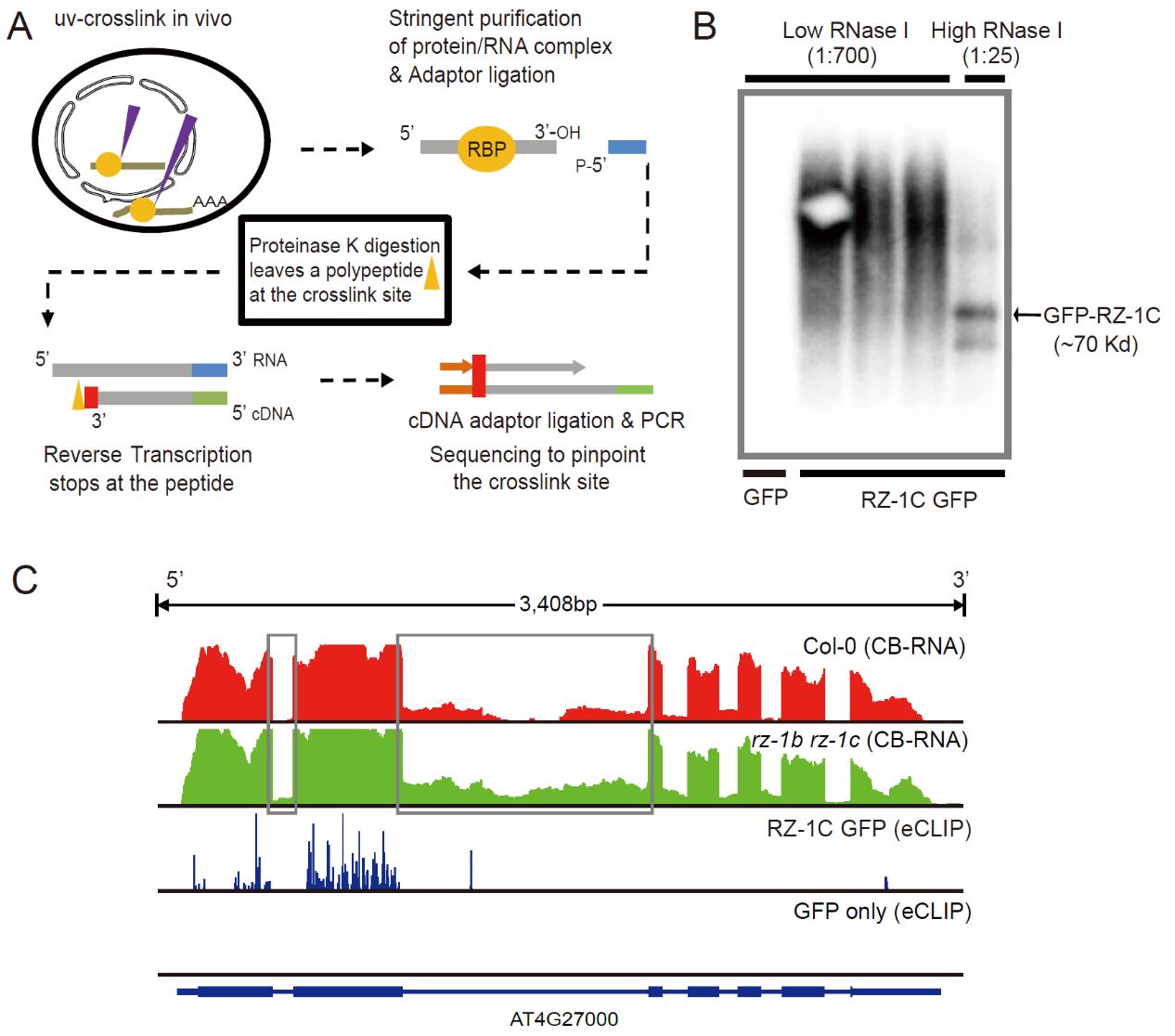
A:示意图展示了eCLIP-seq的原理及实验流程;B:eCLIP-seq实验中捕捉到的被RZ-1C蛋白结合的RNA放射自显影图像;C: 一个共转录剪接受到RZ-1C直接调控的基因实例。图片显示了在该基因上的CB-RNA-seq结果及eCLIP-seq结果
在方法学上,研究团队首次在植物中建立了两种非常有价值的实验技术体系。第一种技术CB-RNA-seq可用于研究正在转录的CB-RNA的分布情况。该技术借助高严谨性的尿素洗涤方法,去除细胞核中游离的RNA和蛋白,富集和染色质高度紧密结合的蛋白及核酸,是一种简单易行的研究新生RNA的实验方法。同时作者给出了新生RNA对应的简单的数学解释,为该实验技术的应用提供了新的视角;第二种技术eCLIP-seq可用于高精度、高严谨性定位RNA结合蛋白结合底物及结合位点,代表了CLIP技术的最新升级版本,前人在动物细胞中已经使用该技术成功定位了200多个RNA结合蛋白的底物。经过改进和优化,研究团队成功的将该技术用于研究植物中的蛋白-RNA互作。在eCLIP的基础上,研究团队加入了同位素标记RNA末端的操作步骤,能更准确地评估蛋白与RNA的结合情况。这两个实验技术体系的建立将对在植物中开展RNA以及转录相关的研究起到积极的促进作用。

课题组合影(吴柘:二排右二;朱丹灵:一排右二)
此项工作由吴柘课题组牵头完成,生物信息学分析部分由吴柘课题组与南京农业大学作物遗传与种质创新国家重点实验室、生物信息中心教授吴玉峰课题组合作完成。吴柘和吴玉峰为文章共同通讯作者;吴柘课题组的研究助理教授朱丹灵与吴玉峰课题组的博士研究生毛飞为论文的共同第一作者。课题的部分前期工作得到了英国约翰英纳斯中心的Caroline Dean教授,北京大学生命科学院的瞿礼嘉教授及顾红雅教授的大力支持。福建农林大学林学中心教授顾连峰参与了该项工作。
该课题得到了国家自然科学基金,广东省科技厅,深圳市科创委以及南科大科研启动经费的支持。
文章链接:
https://www.sciencedirect.com/science/article/pii/S1674205219303685
供稿:生物系吴柘课题组
最新动态
-
生命科学学院“发现绿美南科”校园动植物科普志愿讲解活动顺利开展
Date:2026-02-24
-
南科大曾福星团队联合Raven H. Huang团队阐明牙龈二氧化碳嗜纤维菌中PrfH识别停滞核糖体的双重机制
Date:2026-02-05
-
筑牢安全屏障,护航科研前行|生命科学学院开展寒假实验室安全检查及系列专项工作
Date:2026-02-03
-
生命科学学院举办“化学与结构生物学科研进展报告交流”总结暨表彰会议
Date:2026-01-09
