南科大王泽峰团队开发深度学习模型 Translation AI,助力揭示RNA翻译调控新机制
日期:2025-05-07
近日,南方科技大学生命科学学院系统生物学系王泽峰讲席教授团队成功开发 Translation AI 模型,在国际期刊 Nucleic Acids Research 发表题为“Analysis of RNA translation with a deep learning architecture provides new insight into translation control”的研究论文。该研究构建了一种基于深度神经网络的模型,可准确预测 RNA 分子的翻译起始位点(Translation Initiation Sites, TIS)和终止位点(Translation Termination Sites, TTS),并揭示了密码子使用偏好在翻译终止过程中的潜在调控作用,为理解转录后调控机制提供了新工具和新思路。
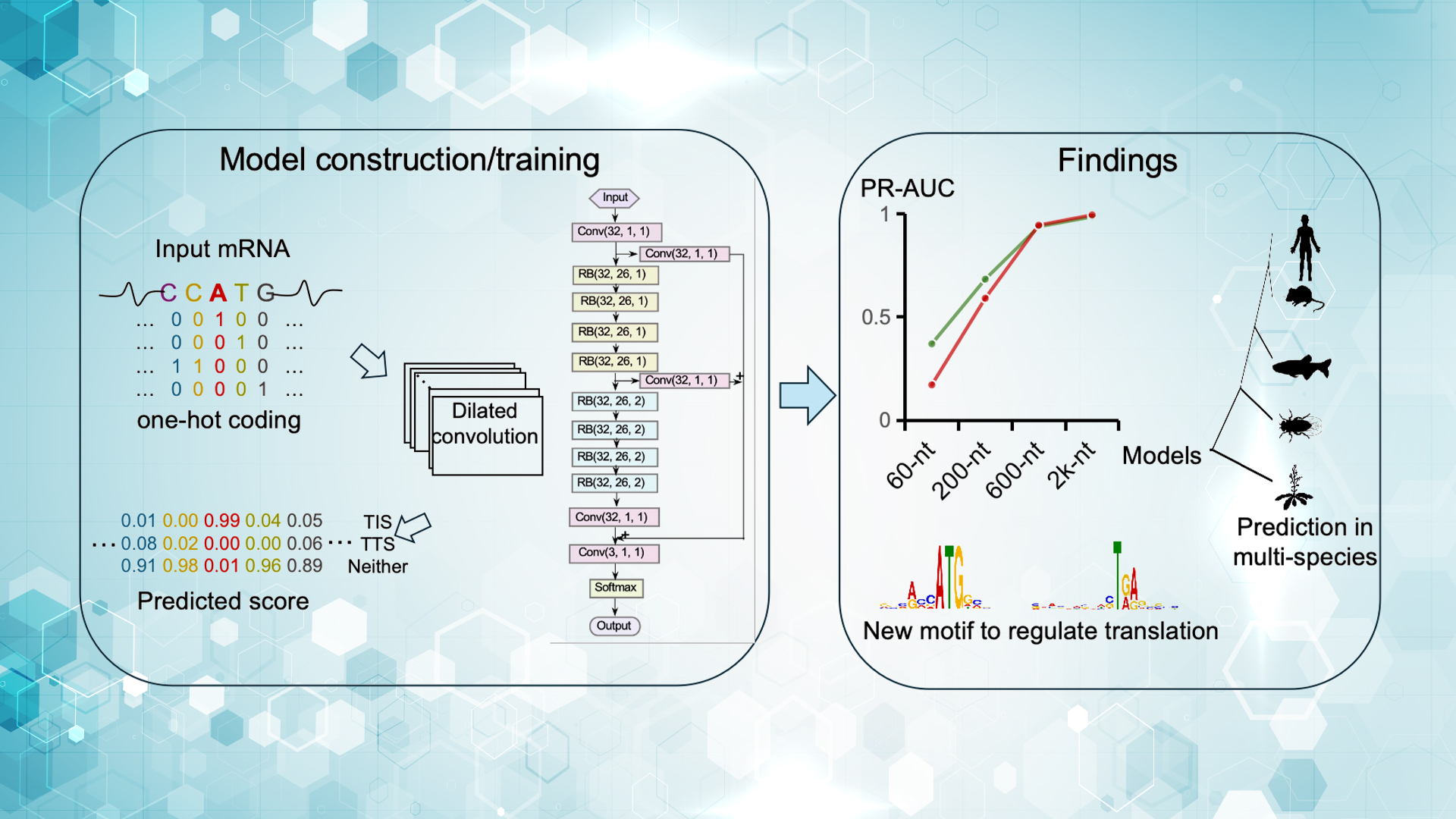
图1 Translation AI 模型架构及研究流程图:模型输入为RNA全长序列,输出为每个位点作为TIS/TTS的
概率;研究结合实验验证进一步探索了RNA翻译的调控机制
在真核细胞中,RNA 分子的翻译起始和终止是高度受调控的生物过程,直接关系到蛋白质的表达与功能异常。然而,现有的基因注释和实验技术在识别翻译位点、预测潜在编码区域等方面仍面临较大挑战。尤其是对于 lncRNA 等非编码 RNA 中可能存在的开放阅读框(ORFs),传统注释方法常常难以捕捉其功能潜力。
为了突破上述瓶颈,研究团队设计并训练了 Translation AI 模型。该模型基于多层级扩张卷积神经网络架构,使用全长 mRNA 序列作为输入,在不依赖已知特征或人工注释的前提下,从序列中学习出翻译调控的隐藏规则。
模型在34,000余条人类参考转录本数据上训练,并在独立测试集上取得了超过0.99的 PR-AUC 指标,性能显著优于现有方法。此外,Translation AI 对模式生物(酵母、拟南芥、斑马鱼等)以及病毒(如SARS-CoV-2与埃博拉病毒)均展现出良好的泛化能力,表明翻译调控机制在物种间具有高度保守性。
Translation AI 不仅能够准确预测翻译位点,更意外揭示了一个全新的调控机制:密码子使用偏好对翻译终止效率具有显著影响。研究人员发现,终止密码子上游的密码子若为C/G含量较高的同义密码子,则翻译终止更为高效;相反,若为A/U富集密码子,则更易发生翻译延伸。该发现通过多种构建的体外报告系统证实,揭示了 mRNA 序列“隐性编码”在翻译控制中的关键作用。
研究还利用 Translation AI 系统性地预测了人类转录组中的潜在新 ORFs,包括673个上游 ORFs(uORFs)、127个下游 ORFs(dORFs)和3794个 lncRNA 中的新 TIS-TTS 对。多个新预测的 ORFs 在蛋白质组数据和功能研究中得到了支持,提示非编码 RNA 可能广泛存在未被发现的编码潜力。
目前,Translation AI 已上线为网页工具,供科研人员上传 RNA 序列并预测其翻译起始与终止位点(网址:https://www.biosino.org/TranslationAI/),以便研究人员分析各类转录本的翻译潜力。
未来,研究团队计划将该模型扩展至非经典翻译位点预测、多物种适配、突变功能注释等领域,推动其在肿瘤转录组、罕见疾病机制及 RNA 药物开发中的应用。
王泽峰和原中国科学院上海营养与健康研究所、现美国国立卫生研究院关节肌肉骨骼及皮肤病研究所樊晓娟博士为论文共同通讯作者,樊晓娟、美国国立卫生研究院癌症研究所常天根和中国科学院上海营养与健康研究所博士研究生陈楚赟为论文共同第一作者。美国国立卫生研究院关节肌肉骨骼及皮肤病研究所 Markus Hafner 博士也参与了研究。本研究得到国家自然科学基金、科技部国家重点研发计划生物大分子与微生物组专项和中国科学院战略性先导科技专项(B 类)等项目的资助。
论文链接:https://doi.org/10.1093/nar/gkaf277
供稿:生命科学学院
通讯员:邹冬霞、罗舒文
主图:丘妍
编辑:曾昱雯
最新动态
-
蕙质兰心·芳华绽放|生科院举办“三八”国际妇女节主题插花活动
Date:2026-03-06
-
生命科学学院“发现绿美南科”校园动植物科普志愿讲解活动顺利开展
Date:2026-02-24
-
南科大曾福星团队联合Raven H. Huang团队阐明牙龈二氧化碳嗜纤维菌中PrfH识别停滞核糖体的双重机制
Date:2026-02-05
-
筑牢安全屏障,护航科研前行|生命科学学院开展寒假实验室安全检查及系列专项工作
Date:2026-02-03
