南科大戴紫薇团队开发代谢网络热力学分析算法dGbyG
日期:2025-09-29
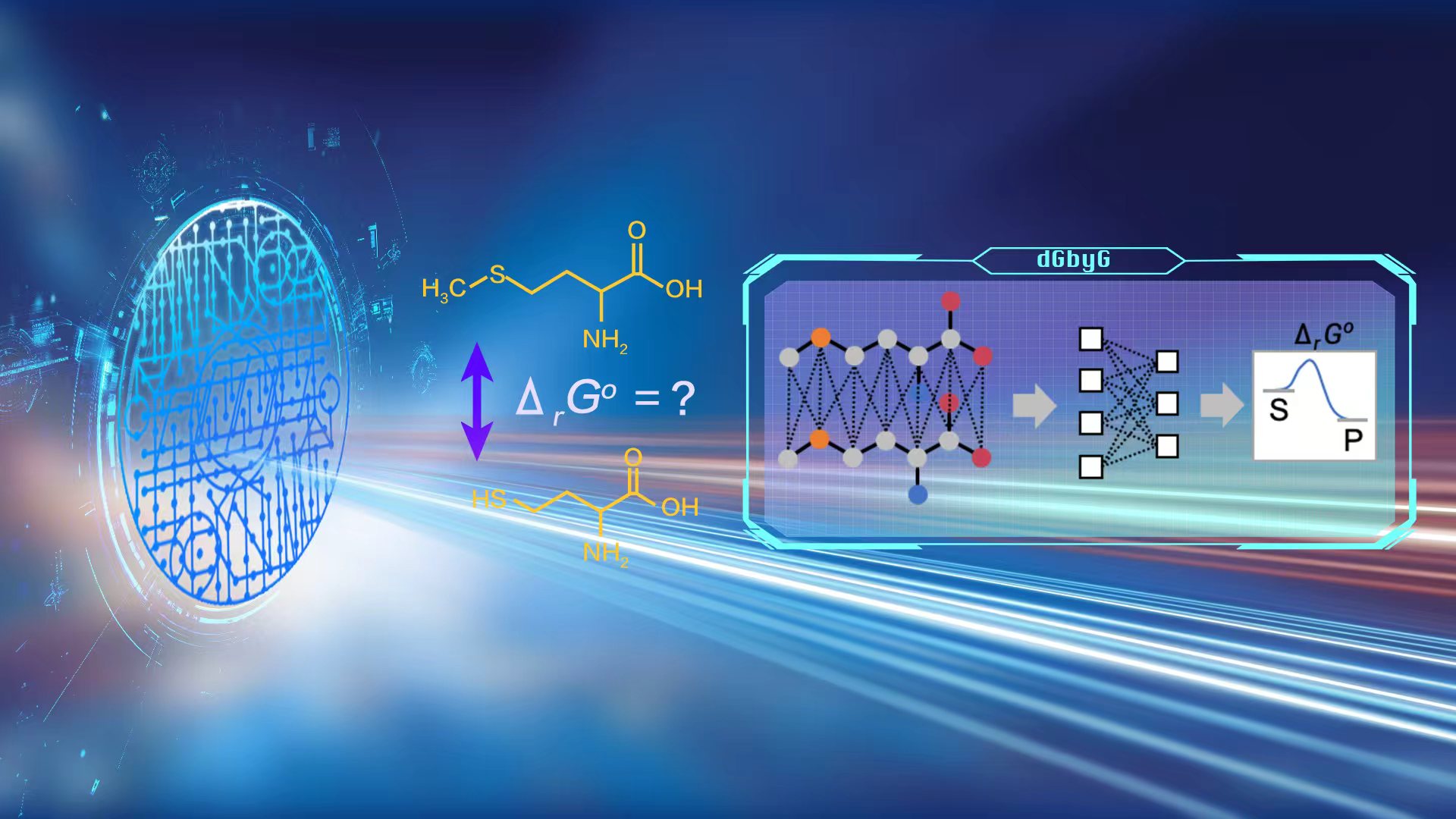
近日,南方科技大学戴紫薇助理教授团队在Cell Press细胞出版社旗下期刊Cell Systems发表题为“Unraveling principles of thermodynamics for genome-scale metabolic networks using graph neural networks”的研究论文。该研究基于图神经网络开发了代谢反应标准吉布斯自由能预测工具dGbyG,利用该工具及热力学流平衡分析,在人类和酵母模型中鉴定出26个错误反应,证实热力学数据对构建高质量代谢网络至关重要。研究进一步揭示了强驱动反应在代谢通路中的拓扑分布规律及其帕累托最优性基础。
“生命以负熵为食”强调了热力学第二定律在生命活动中的重要作用。在宏观层面上,热力学定律约束了生命过程的可能性,而在新陈代谢过程中,热力学定律也决定了生理条件下代谢反应发生的方向,以及代谢通路的调控模式。代谢通路的控制模式与通路中代谢反应的吉布斯自由能分配高度相关,例如在糖酵解通路中,关键调控酶HK、PFK和PK催化的反应具有较负的吉布斯自由能变,从而成为整个通路的关键限速步骤。21世纪以来,随着高通量测序和全基因组代谢网络建模方法的发展,全基因组代谢网络(GEM)已经成为代谢研究的标准技术工具。一个物种的GEM通常包括数以千计的代谢反应,但现有实验数据和预测方法仅能提供少数代谢反应的标准吉布斯自由能,使得在全基因组水平上研究代谢网络的热力学特性和控制模式极为困难。

本研究基于图神经网络开发了代谢反应标准吉布斯自由能预测工具——dGbyG,将验证集预测误差的中位数由现有最优方法的5.33 kJ/mol减小到4.11 kJ/mol,并对人类全基因组代谢网络模型Recon3D中反应的覆盖度由64.14%提升到71.22%,达到了基于分子结构的预测方法的极限。相比传统的反应热力学预测算法——基团贡献法而言,dGbyG无需依赖预定义的化学基团,而是直接对分子中的原子和键进行建模,使得它具有强大的稳健性和泛化能力,在训练数据稀缺时仍然能够保持优越的预测精度,并且对训练集中未出现的反应机制,也可以作出准确的预测。
热力学的基本原理根据每个代谢反应的吉布斯自由能变,从两个方面决定了代谢网络的行为。代谢反应吉布斯的自由能变是一个同时由标准吉布斯自由能变和代谢物浓度决定的热力学量,它的符号指示了反应可以自发进行的方向,而它的数值大小决定了重要限速步骤的分布。因此,dGbyG所产生的高精度反应热力学数据,可以从这两方面深化我们对代谢网络设计原理的理解。研究团队首先利用dGbyG和热力学流平衡分析(TFBA)算法,在人类和酵母的3个全基因组代谢网络中鉴定出26个错误的反应,证明准确反应热力学数据对于构建高质量的全基因组代谢网络不可或缺。
最后,本研究系统分析了人类代谢反应热力学性质的一般规律,发现网络中的强热力学驱动反应具有特殊的拓扑结构特征,倾向于分布在通路的两端。代谢在进化中受到多方面的选择压力,包括效率、可控性、成本、结构复杂性等,这些因素共同决定了具有何种结构和动力学行为的代谢网络,会在进化过程中被选择。为了对强热力学驱动反应的聚集模式进行解释,本研究对代谢效率、酶成本和代谢物成本之间的权衡进行了理论建模,结果表明这三者在达到最优平衡时,强热力学驱动反应分布在通路两端。理论结果和真实代谢网络热力学模式的一致性,表明效率优化和成本节约的经济学法则在代谢网络的进化中起到重要作用。
南方科技大学生命科学学院硕士生范文超(现为北大-清华生命科学联合中心博士生)为论文第一作者,戴紫薇为论文通讯作者。南方科技大学为论文第一单位。研究得到了国家自然科学基金委、科技部重点研发计划、广东省科技厅、深圳市科技创新委员会的资金支持。
文章链接:https://www.cell.com/cell-systems/abstract/S2405-4712(25)00226-1
供稿:生命科学学院
文字:戴紫薇
通讯员:李沐涵
主图:丘妍
最新动态
-
南科大曾福星团队联合Raven H. Huang团队阐明牙龈二氧化碳嗜纤维菌中PrfH识别停滞核糖体的双重机制
Date:2026-02-05
-
筑牢安全屏障,护航科研前行|生命科学学院开展寒假实验室安全检查及系列专项工作
Date:2026-02-03
-
生命科学学院举办“化学与结构生物学科研进展报告交流”总结暨表彰会议
Date:2026-01-09
-
生命科学学院党委召开2025年度党组织书记抓基层党建工作述职评议会议
Date:2026-01-08
