生科院陈炜课题组开发一种全新的单细胞测序混样技术
日期:2021-04-06
近日,南方科技大学生命科学学院陈炜课题组在Molecular Systems Biology上发表了CASB: A concanavalin A-based sample barcoding strategy for single-cell sequencing的研究,报道了一种名为CASB的新型单细胞测序样本混样技术。

单细胞测序(包括scRNA-seq和snATAC-seq等)已经成为研究多细胞样本转录组和表观遗传组异质性的强大工具。早期的单细胞测序流程仅限一次分析数十到数百个单个细胞。随着微滴技术(droplet)[1, 2]、微孔技术(microwell)[3]和组合索引技术(combinatorial index)[4, 5]的引入,现在的单细胞测序技术可同时对数千、上万个单细胞或细胞核进行分析。单细胞测序通量的增加不仅降低了每个细胞的试剂成本,而且使得利用单细胞测序对整个器官或整个生物个体进行分析成为可能。此外,单细胞测序也逐渐被用于研究具有异质性细胞群体对不同刺激在不同时间尺度上的响应,需要对十几种或几十种样本同时进行单细胞测序分析。
在常规的单细胞实验中,每个单细胞样本文库特有的条形码(barcode)是在文库构建的最后一步才被引入。这就要求在单细胞测序实验中,平行地、单独地对多个样本进行处理。这不仅增加了测序成本和工作量,还限制了样本的分析通量。为了克服这个问题,多种允许在单细胞测序实验之前对样本进行混样的技术被开发出来。在这些技术中,每个样品中的细胞被一种独特的barcode标记。在单细胞测序文库制备过程中,样品特异的barcode能够与细胞barcode进行连接,最终实现样本的拆分。这些技术主要分为两大类,依赖于遗传机制和不依赖于遗传机制。研究人员可利用遗传机制使细胞表达一个外源基因,该基因的3'UTR带有样本特异的barcode,可像内源基因一样在单细胞测序中被捕获,最终使得样品特异的barcode能够与细胞barcode进行连接[6, 7];但该类技术的应用对象比较有限。在不依赖于遗传机制的技术中(总结于下表),研究人员利用不同的手段将带有样本特异barcode的核酸序列铆定在细胞或细胞核表面,使得该barcode能够在单细胞测序中被捕获[8-11]。但是,现有的技术均有各自的局限性,其中包括可延展性(scalability)、通用性(universality)和引入人为干扰的可能性等方面。而且,所有这些方法仅能与scRNA-seq配合使用,与snATAC-seq不兼容。
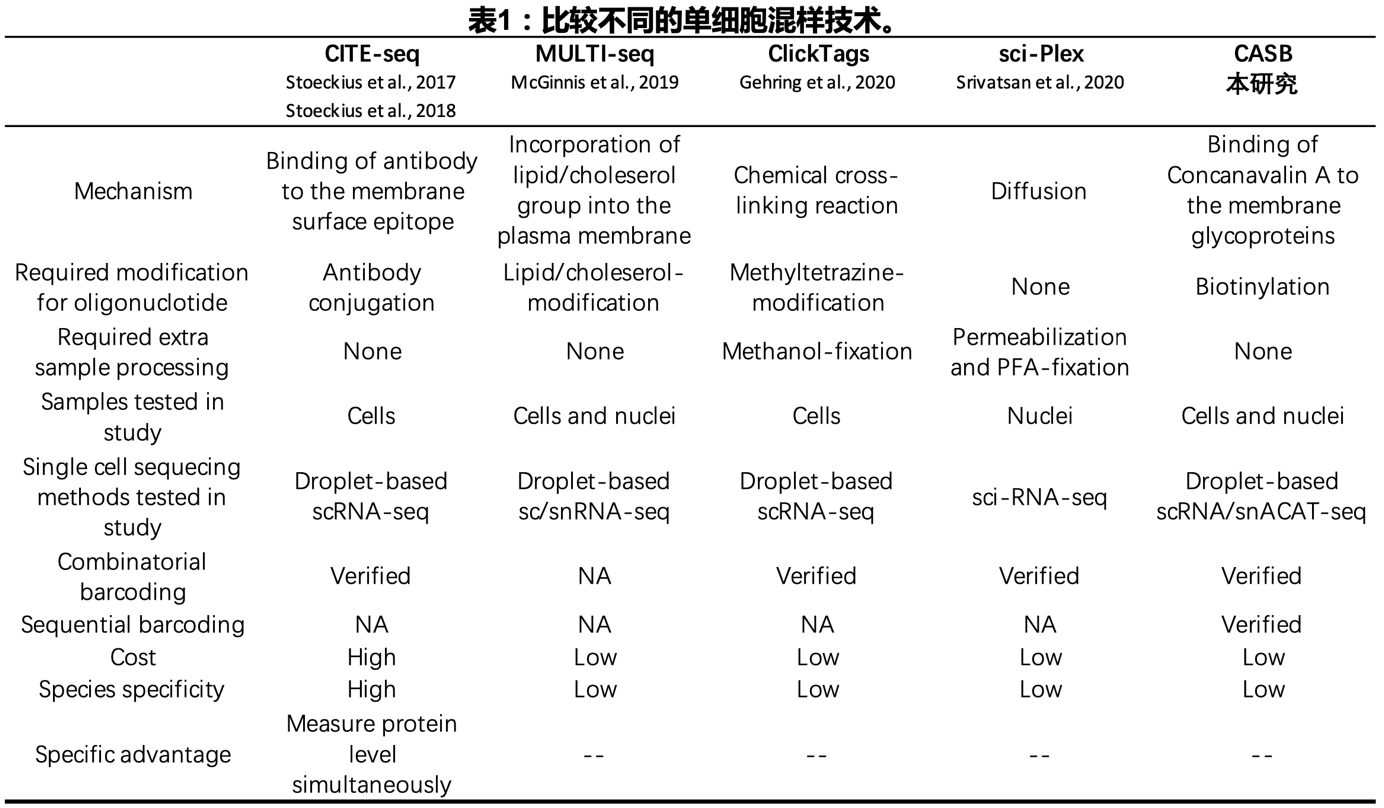
陈炜课题组开发的单细胞测序混样技术,克服了许多现有技术的局限性,利用伴刀豆球蛋白A(ConA)的糖蛋白结合能力,通过链霉亲和素桥连生物素化的单链DNA(带有样本特异barcode的核酸序列)标记细胞或细胞核。CASB可以完美适配scRNA-seq和snATAC-seq的工作流程,对样本的遗传背景没有偏好,能够高效、准确地实现样本的拆分,并帮助鉴定细胞双胞(cell doublets)。
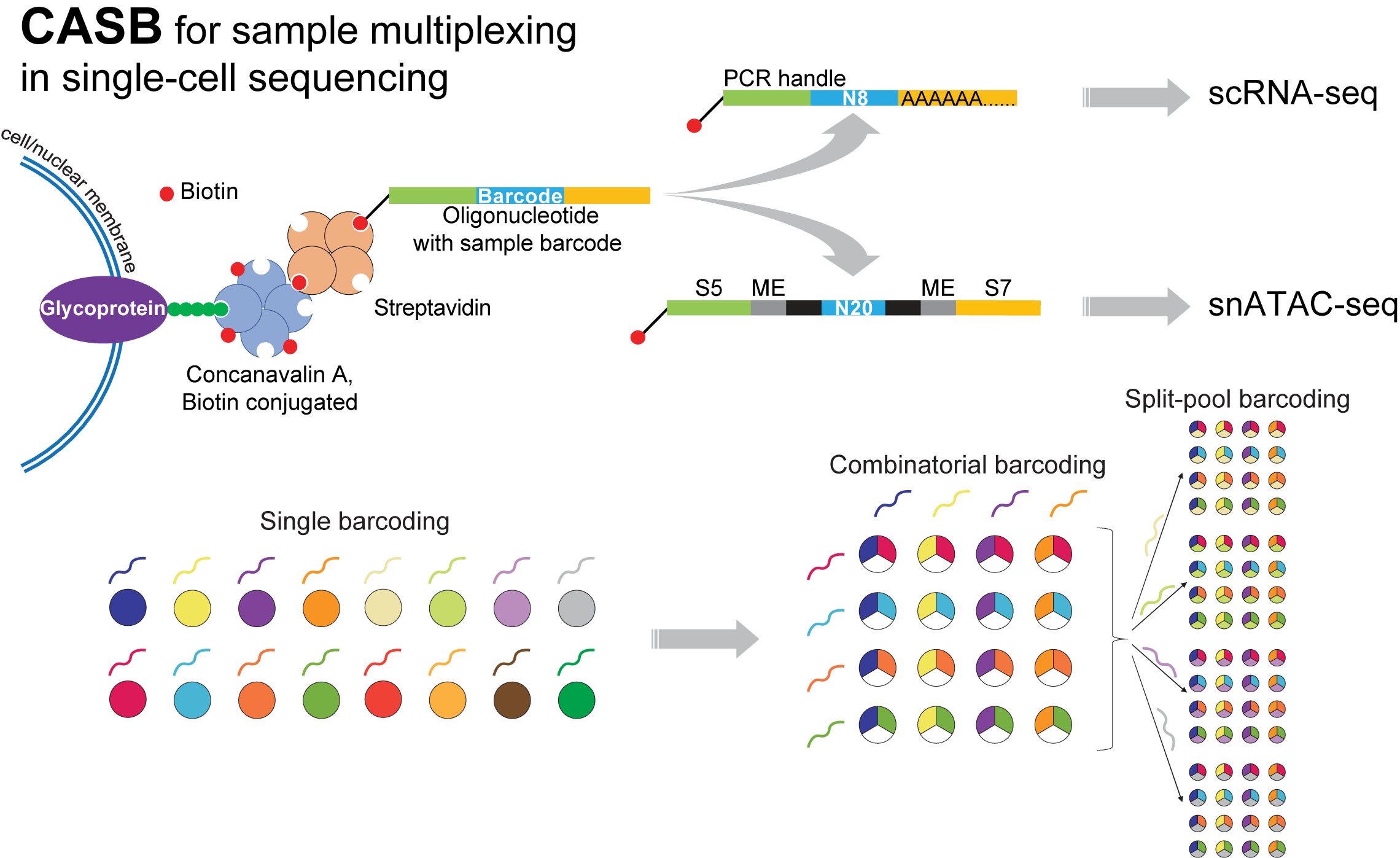
在该研究中,作者结合CASB和scRNA-seq对乳腺细胞MDA-MB-231在不用药物处理后不同时间点的转录组进行了分析,揭示了该细胞内部的异质性和不同细胞亚群对药物反应的异质性,并提示潜在的耐药机制。作者将CASB应用于scRNA-seq和snATAC-seq,描绘了IFN-γ介导的表观基因组和转录组图谱的动态变化,鉴定出介导IFN-γ反应异质性的转录因子。最后,作者将组合索引应用于CASB标记,展示了CASB技术极高的延展性。随着单细胞测序通量的不断提高,CASB技术可以进一步降低实验成本,改善数据分析,最大程度地减少批次差异,而且还可以整合更多类型的信息,包括时间、处理条件、甚至空间坐标,有望成为该领域的重要工具。
南方科技大学生命科学学院生物系讲席教授陈炜、副研究员方亮为本文的共同通讯作者,方亮、副研究员李贵鹏为本文的并列第一作者。该研究得到了深圳市基因调控与系统生物学重点实验室、深港脑科学创新研究院、国家自然科学基金的资助。
文章链接:https://www.embopress.org/doi/full/10.15252/msb.202010060
陈炜课题组链接:http://chenlab.bio.sustech.edu.cn/
供稿:陈炜课题组
文字、图片:方亮
通讯员:付文卿
参考文献:
1. Macosko, E.Z., et al., Highly Parallel Genome-wide Expression Profiling of Individual Cells Using Nanoliter Droplets. Cell, 2015. 161(5): p. 1202-1214.
2. Klein, A.M., et al., Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell, 2015. 161(5): p. 1187-1201.
3. Gierahn, T.M., et al., Seq-Well: portable, low-cost RNA sequencing of single cells at high throughput. Nat Methods, 2017. 14(4): p. 395-398.
4. Cusanovich, D.A., et al., Multiplex single cell profiling of chromatin accessibility by combinatorial cellular indexing. Science, 2015. 348(6237): p. 910-4.
5. Cao, J., et al., Comprehensive single-cell transcriptional profiling of a multicellular organism. Science, 2017. 357(6352): p. 661-667.
6. Hurley, K., et al., Reconstructed Single-Cell Fate Trajectories Define Lineage Plasticity Windows during Differentiation of Human PSC-Derived Distal Lung Progenitors. Cell Stem Cell, 2020. 26(4): p. 593-608 e8.
7. Weinreb, C., et al., Lineage tracing on transcriptional landscapes links state to fate during differentiation. Science, 2020. 367(6479).
8. McGinnis, C.S., et al., MULTI-seq: sample multiplexing for single-cell RNA sequencing using lipid-tagged indices. Nat Methods, 2019. 16(7): p. 619-626.
9. Stoeckius, M., et al., Simultaneous epitope and transcriptome measurement in single cells. Nat Methods, 2017. 14(9): p. 865-868.
10. Gehring, J., et al., Highly multiplexed single-cell RNA-seq by DNA oligonucleotide tagging of cellular proteins. Nat Biotechnol, 2020. 38(1): p. 35-38.
11. Srivatsan, S.R., et al., Massively multiplex chemical transcriptomics at single-cell resolution. Science, 2020. 367(6473): p. 45-51.
最新动态
-
蕙质兰心·芳华绽放|生科院举办“三八”国际妇女节主题插花活动
Date:2026-03-06
-
生命科学学院“发现绿美南科”校园动植物科普志愿讲解活动顺利开展
Date:2026-02-24
-
南科大曾福星团队联合Raven H. Huang团队阐明牙龈二氧化碳嗜纤维菌中PrfH识别停滞核糖体的双重机制
Date:2026-02-05
-
筑牢安全屏障,护航科研前行|生命科学学院开展寒假实验室安全检查及系列专项工作
Date:2026-02-03
