南科大生科院翟继先团队合作揭示水稻non-CG甲基化调控模式
日期:2021-06-15
近日,南方科技大学生命科学学院生物系副教授翟继先团队与中国农业科学院水稻研究所王克剑团队合作,在The Plant Cell在线发表题为“Multiplex CRISPR-Cas9 editing of DNA methyltransferases in rice uncovers a class of non-CG methylation specific for GC-rich regions”的研究论文。
真核生物基因组中广泛存在DNA胞嘧啶(C)甲基化修饰。绝大多数动物DNA甲基化发生在CG(也称作CpG)序列的C位点,而植物DNA甲基化可以发生在CG序列和non-CG序列(包括CHG和CHH)的C位点,DNA甲基化是由DNA甲基转移酶所维持的。水稻non-CG甲基化缺失会出现比拟南芥更为明显的生长发育缺陷,因此揭示水稻non-CG甲基化的调控模式具有重要意义。目前仅有少数水稻non-CG甲基转移酶被研究过,对水稻的non-CG甲基化调控模式还知之甚少。
团队研究系统解析了水稻中所有non-CG DNA甲基转移酶的功能,发现了水稻中有一类特殊的富含GC序列的区域的non-CG甲基化在主效non-CG甲基转移酶(CMT2,CMT3和DRM2)缺失的情况下仍然不受影响,作者进一步研究发现这些区域的non-CG甲基化可以由OsCMT3b维持。
团队研究利用了一种适用于水稻的高效CRISPR-Cas9多基因编辑技术,获得了水稻7个non-CG甲基转移酶的单基因突变体,以及它们的组合突变。全基因组甲基化测序(WGBS)分析表明:OsCMT2, OsCMT3a和OsDRM2是水稻中主效non-CG DNA甲基转移酶,它们的缺失会导致基因组大量区域的CHG和CHH甲基化水平降低,而其它non-CG DNA甲基转移酶的缺失不会导致明显的non-CG甲基化水平变化。当同时敲除主效的non-CG DNA甲基转移酶(Os-dcc突变体)后,发现基因组中还有一部分区域维持non-CG甲基化,这些区域具有富含GC的特征。而这些Os-dcc突变体中剩余的甲基化位点在Os-ddccc(Osdrm2/drm3/cmt2/cmt3a/cmt3b)突变体中表现出了明显的non-CG甲基化的丢失。进一步研究发现,这些剩余甲基化位点在Os-ddcc(Osdrm2/drm3/cmt2/cmt3a)突变体中仍然得到维持,具有和Os-dcc突变体一致的non-CG甲基化水平。以上结果表明,OsCMT3b维持了水稻中一类特殊的富含GC序列区域的non-CG甲基化。OsCMT3b是水稻特有的non-CG DNA甲基转移酶,揭示了水稻独特的DNA甲基化调控系统。
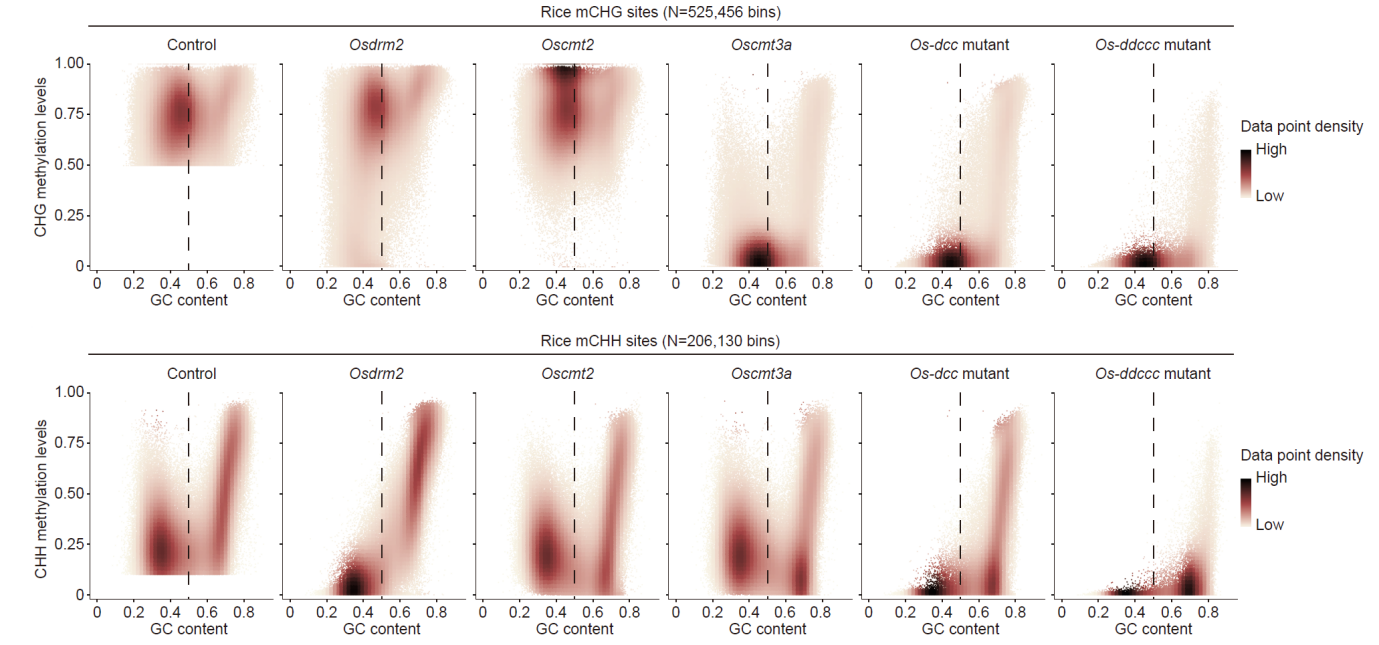
各突变体中,CHG/CHH 甲基化位点的non-CG甲基化水平随GC含量的变化趋势
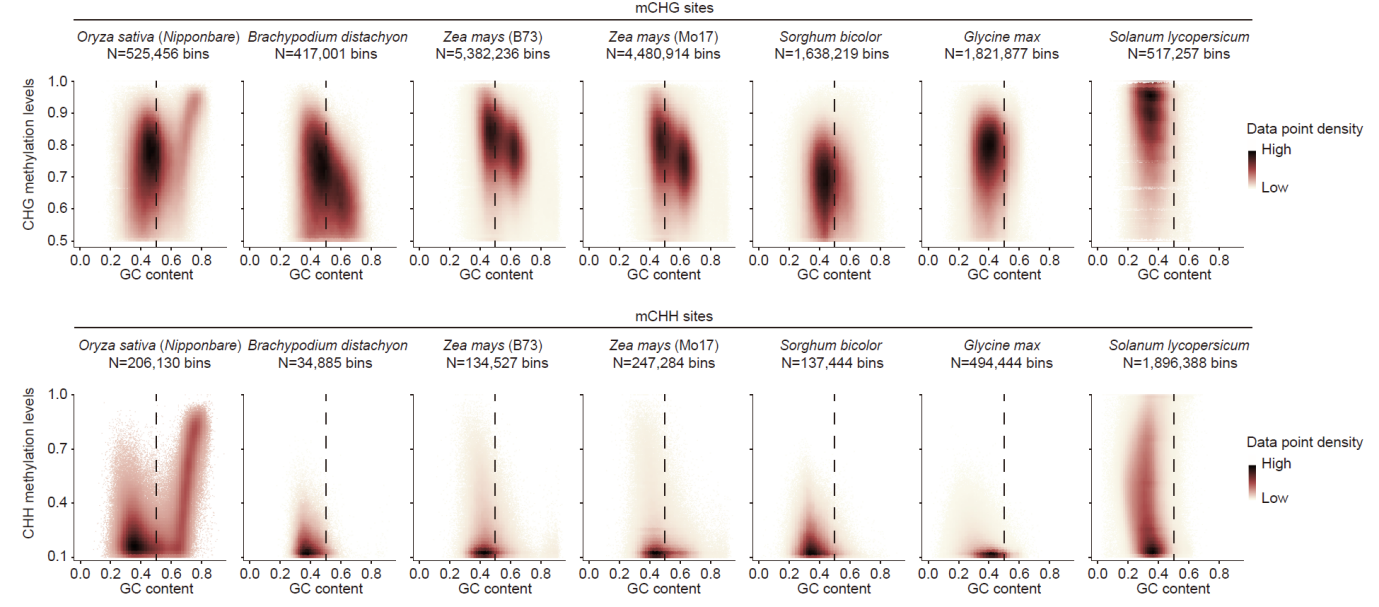
不同植物中,CHG/CHH 甲基化位点的non-CG甲基化水平随GC含量的变化趋势
翟继先与王克剑为论文的共同通讯作者,南科大博士后胡道恒、博士生于义溟和中国农科院学者王春为论文共同第一作者。南科大为第一单位。美国威斯康星大学麦迪逊分校教授Xuehua Zhong,生物系教授杜嘉木,东北师范大学教授宫磊和华南农业大学教授夏瑞参与了该项研究。该研究得到了基金委,广东省创新创业团队,深圳市科创委, 中国农科院创新工程等项目的资助。
原文链接:https://doi.org/10.1093/plcell/koab162
供稿:生命科学学院
文字:胡道恒、于义溟
图片:翟继先课题组
通讯员:付文卿
编辑:劳湘雯
最新动态
-
生科院五周年|本科生学科竞赛与论文发表交流分享会举行
Date:2025-12-12
-
生科院五周年|“创新挑战班”三课联动:班会凝共识、院长面对面、校友传薪火
Date:2025-12-12
-
南科大生科院“五载求索·创生未来”学术研讨会成功举办,擘画生命科学发展新蓝图
Date:2025-12-11
-
校企合作再进阶,华润-南科大生命健康研究院转化研讨会成功举办
Date:2025-11-28
